高通量、自动化单细胞力谱测定!多功能单细胞显微操作全新技术助力单细胞力学研究
发布日期:2023-02-22
单程细胞具有复杂生物学性质,它们通过细胞外基质ECM形成紧密的细胞与基质细胞与细胞连接,诸如上皮细胞通过这种特殊的链接方式构成了屏障层保护人体免受外界损伤。因此细胞之间以及细胞基底的粘附力测定对于研究细胞粘附蛋白的机制有着重要意义。使用力学工具测量细胞间以及细胞与基质之间的粘附力始终不是一件容易的事情。首先,由于细胞与基质的作用力仅为nN级别,因此需要力学精度较高的设备才能够测量,而且在这其中较为适合的工具为原子力显微镜(AFM)。原子力显微镜能够提供纳米级别的操作精度并可测量从pN~nN范围的力谱。但是受制于AFM探针本身的限制,需要借助修饰手段才能够让细胞与探针固定到一起,这个过程十分繁琐,并且由于需要大量手工操作很难实现高通量的测量。而不同的细胞由于细胞异质性使得要想确定粘附力需要较多样本才能获得相对准确的值,无法实现高通量测量直接限制了原子力探针在细胞粘附力上的应用。
而多功能单细胞显微操作FluidFM技术的出现改变了这一现状,它使用特殊的中空探针能够轻松地通过负压抓取细胞,取得和AFM近似精度的数据,无需在探针上进行任何修饰,不会改变细胞表面的任何通路,从而能够得到接近细胞原生的数据。在实验结束后能够通过正压快速丢弃用过的细胞,具备很高的自动化,能够快速测量细胞粘附力。
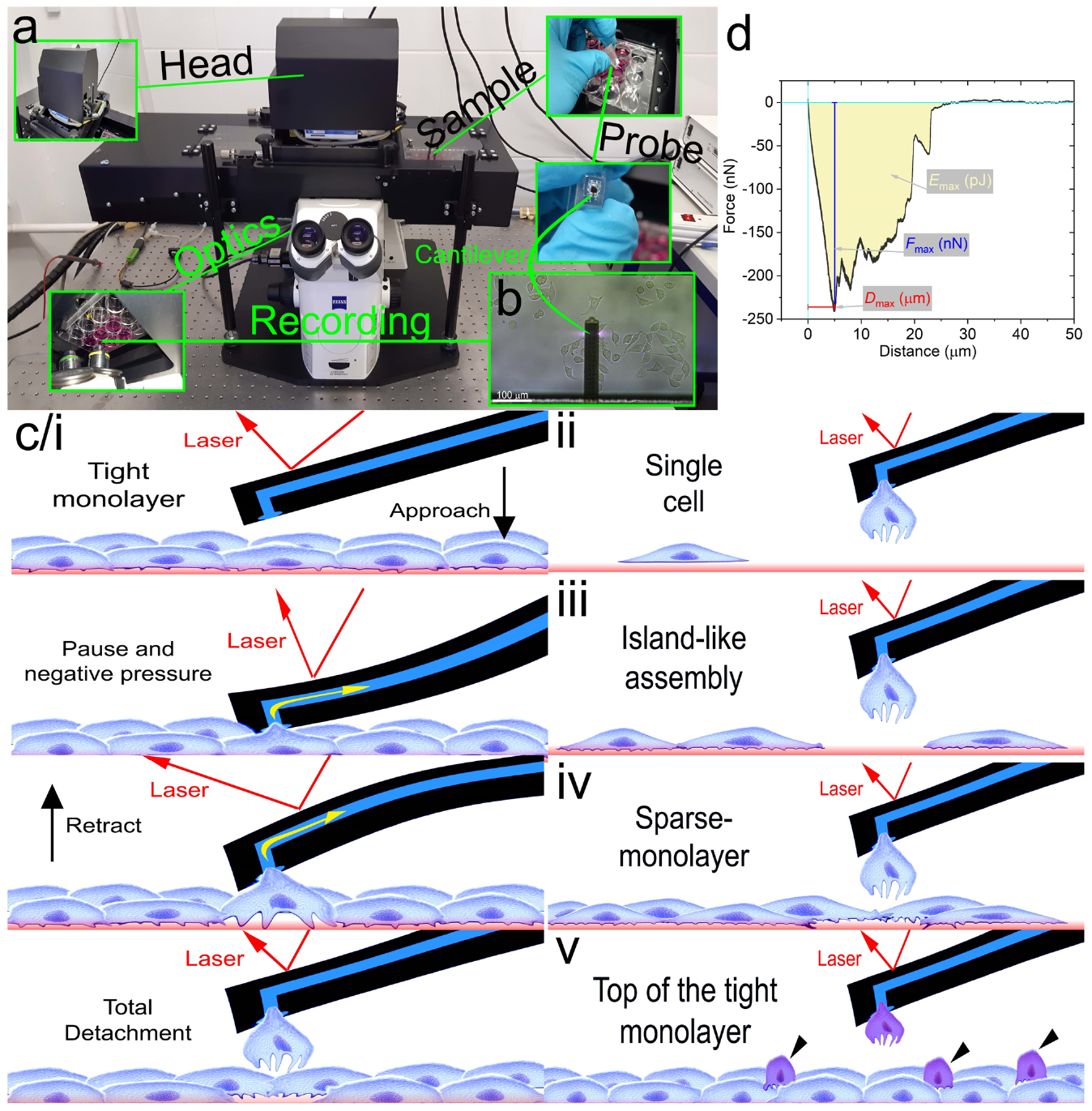
使用FluidFM对细胞操作的基本流程
FluidFM在粘附力测量上具备显著优势。如图所示,FluidFM能够通过负压将细胞吸附到原子力探针的末端,通过高精度位移台的控制将细胞从基底上分离,并且同时记录FD曲线。通过FD曲线能够获得最大粘附力Fmax和粘附能量Emax。通过高度自动化的控制系统能够在短时间内测量大量细胞粘附力,评估细胞群体分布以及细胞间差异,并且可有效避免传统粘附力测量因准备时间过长而错过最佳测量时间导致的细胞粘附力改变,得到更为精准的结果。近期,Agoston等人使用多功能单细胞显微操作系统FluidFM实现了高通量细胞粘附力测量,对同种细胞不同区以及不同细胞之间的粘附力进行测量和比较。
作者首先对Vero和Hela细胞在不同状态下的粘附力进行了测量和比较,总共测量了214个细胞。通过比较明胶涂层上处于单个细胞、孤岛状细胞、致密连接细胞以及单层细胞上游离细胞之间的粘附力,能够明显观测到Vero细胞处于致密连接的细胞粘附力最大,大概在750 nN左右,随着细胞单细胞层的稀疏,细胞粘附力有所下降,而处于细胞层顶部的细胞粘附力最低仅为50 nN左右。这一点充分说明上皮细胞能够在细胞之间形成紧密的连接,而处于细胞层外的细胞则几乎没有粘附力。而对于HeLa这样的肿瘤细胞测量的结果却显示出了截然不同的结果,处于不同状态的细胞有着近似的粘附力,基本都在200 nN左右,这与处于单个游离上皮细胞的粘附力十分接近,表明HeLa细胞在不同环境下仍然具有较高迁徙能力。
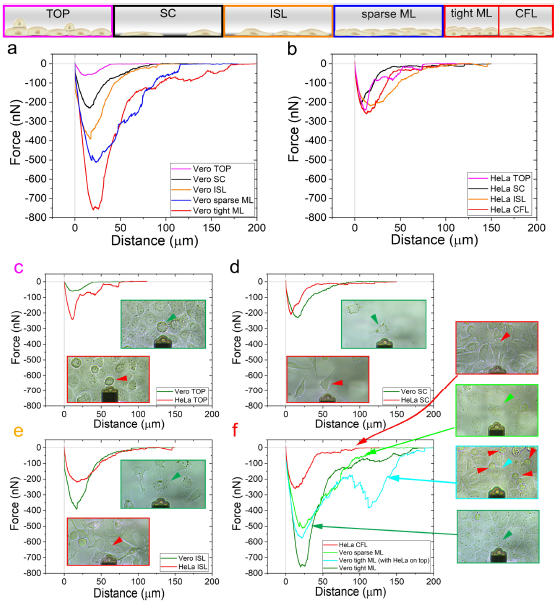
使用FluidFM对不同区域细胞的FD曲线测定结果和对比
通过对这两种细胞的最大粘附力、最大粘附能量、最大拉伸距离和细胞接触面积进行统计分析可以发现,HeLa肿瘤细胞在粘附力和粘附能量上均有所降低,但是当HeLa细胞形成了单层后,两者区别不大。
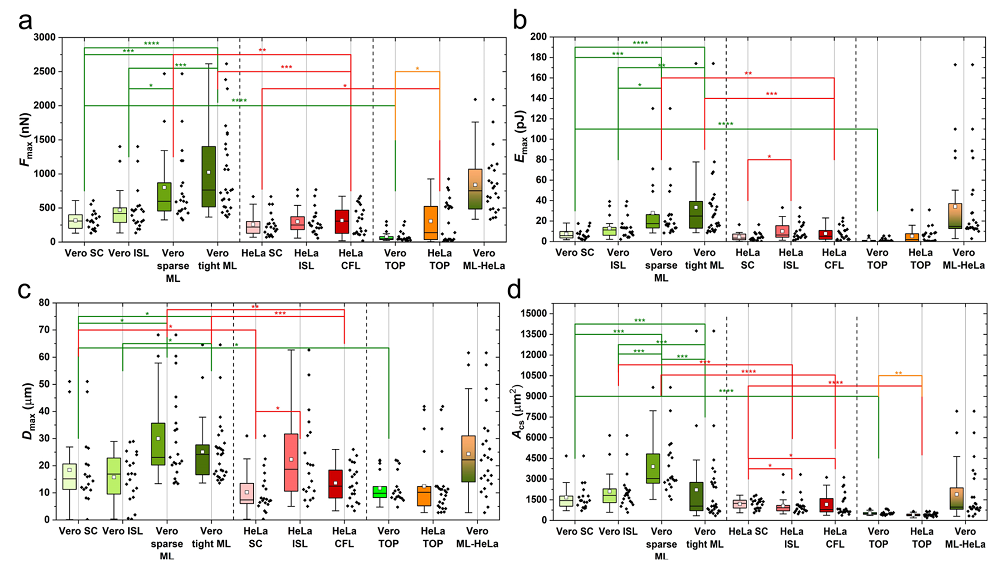
对比Hela和Vero在不同生长状态下的最大粘附力、最大粘附能量、粘附拉伸距离和粘附面积。
再进一步对Vero与HeLa细胞最大粘附力与距离和接触面积进行对比,依然可以得到与单独比较粘附力相同的结果,并且最大能量与细胞接触面积的比值中也存在着类似的结果。由此可见肿瘤细胞通过降低自身粘附力从而获得了更好的迁移能力。
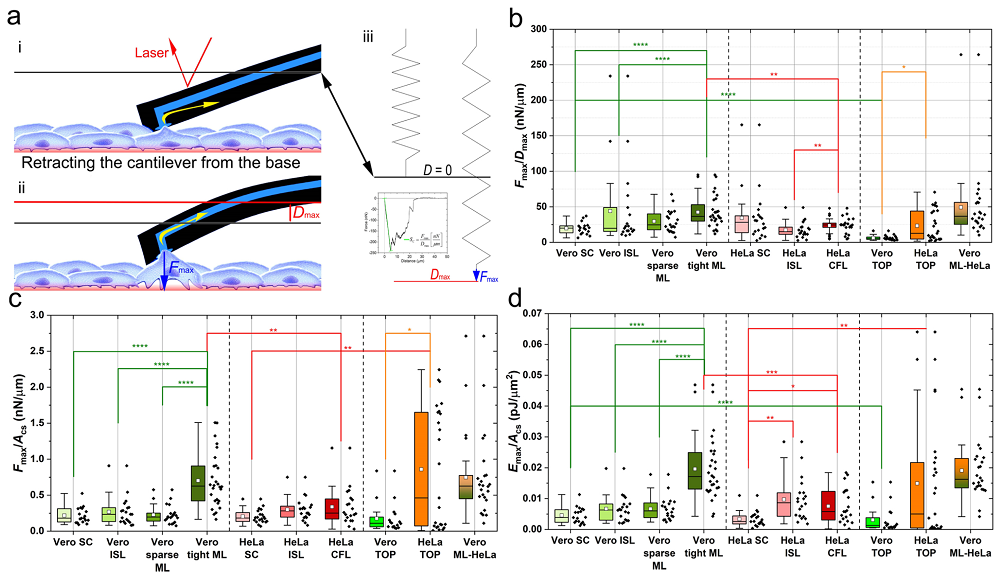
对不同状态Vero和A549之间的粘附力/粘附距离、粘附力/粘附面积、粘附能量/粘附面积
总结
细胞粘附力测定在细胞生命科学研究中起着至关重要的作用,然而传统手段中有着各种各样的局限性,主要原因是缺乏一种有效抓取细胞并进行力学测定的手段。现如今FluidFM技术在细胞粘附力测定中的应用,使得研究者们有了一种能够有效、低损的方式抓取细胞,配合原子力显微镜精确测量的特性,真正意义上做到精准、无损、快速的测量单细胞粘附力,帮助研究者寻找细胞粘附力与细胞生命发展、肿瘤细胞转移之间的关系。
【参考文献】
[1] A. Sancho, M. B. Taskin, L. Wistlich, P. Stahlhut, K. Wittmann, A. Rossi & J. Groll. Cell Adhesion Assessment Reveals a Higher Force per Contact Area on Fibrous Structures Compared to Flat Surfaces. ACS Biomater. Sci. Eng. 2022, 8, 2, 649–658.
[2] P.W. Doll, K. Doll, A. Winkel, R. Thelen, R. Ahrens, M. Stiesch & A.E. Guber. Influence of the Available Surface Area and Cell Elasticity on Bacterial Adhesion Forces on Highly Ordered Silicon Nanopillars. ACS Omega. 2022, 7, 21, 17620–17631.
[3] Sankaran, S. Jaatinen, L. Brinkmann, J. Zambelli, T. Vörös, J. Jonkheijm, P. Cell adhesion on dynamic supramolecular surfaces probed by fluid force microscopy-based single-cell force spectroscopy. ACS Nano 2017, 11, 3867–3874.
[4] Sancho, A. Vandersmissen, I. Craps, S. Luttun, A. Groll, J. A new strategy to measure intercellular adhesion forces in mature cell-cell contacts. Sci. Rep. 2017, 7, 46152.
[5] Ines, Lüchtefeld. Alice, Bartolozzi. Julián M. M. Oana, Dobre. Michele, Basso. Tomaso, Zambelli. Massimo, Vassalli. Elasticity spectra as a tool to investigate actin cortex mechanics. J Nanobiotechnol. 2020, 18, 147.
[6] Dehullu, J. Valotteau, C. Herman-Bausier, P. Garcia-Sherman, M. Mittelviefhaus, M. Vorholt, J. A. Lipke, P. N. Dufrene, Y. F. Fluidic force microscopy demonstrates that homophilic adhesion by Candida albicans Als proteins is mediated by amyloid bonds between cells. Nano Lett. 2019, 19, 3846–3853.
[7] Mittelviefhaus, M. Müller, D. B. Zambelli, T. Vorholt, J. A. A modular atomic force microscopy approach reveals a large range of hydrophobic adhesion forces among bacterial members of the leaf microbiota. ISME J. 2019, 13, 1878–1882.
[8] F. Weigl, C. Blum, A. Sancho & J. Groll. Correlative Analysis of Intra- versus Extracellular Cell Detachment Events vis the Alignment of Optical Imaging and Detachment Force Quantification. Adv. Mater. Technol. 2022, 2200195.


